Proteinsynthese Bakterien - Translation
Nach der Transkription läuft die molekulare Proteinsynthese – Maschinerie an. Bei Bakterien beginnen die Ribosomen noch während der mRNA-Synthese mit der Proteinsynthese, so werden bei E. coli 16 Aminosäuren pro Sekunde verknüpft.
Worum gehts?
Es werden vertiefend die drei Phasen der Translationsvorgänge bei Bakterien besprochen.
In den Phasen sind verschiedene Hilfs-Proteine beteiligt, die kurz vorgestellt werden und deren Wirkungsweise veranschaulicht wird.
Die drei Phasen der Translation
Wie bei der Transkription läuft auch die Translation in drei Schritten ab:
- Bei der Initiation wird das Ribosom zusammengebaut und die erste tRNA wird komplementär am Start-Codon gebunden.
- Die Elongation ist der schrittweise Ablesevorgang, bei dem sich fortlaufen beladene tRNA-Moleküle komplementär anlagern und die Aminosäuren zu einem Peptid verknüpft werden.
- Bei der Termination kommt es zum Abbruch des Ablesevorgangs. Proteinsynthese wird angehalten.
Initiation - der Proteinsynthesestart
Bei der bakteriellen Translation sind drei kleine Hilfs-Proteine, die sogenannten Initiationsfaktoren (IF-1, IF-2 und IF-3), beteiligt.
Die tRNA, die sich am Startcodon anlagert, unterscheidet sich von den nachfolgend tRNA´s. Sie kommt nur bei der Initiation zum Einsatz und wird deshalb als Initiator-tRNA bezeichnet. Aufgrund ihrer chemischen Eigenschaften ist sie als Einzige in der Lage, sich an der P-Stelle anzulagern.
![]() Die Ribosomenbindungsstelle - Shine - Dalgarno - Sequenz
Die Ribosomenbindungsstelle - Shine - Dalgarno - Sequenz
Wie findet das Ribosom den Ort einer exakten Anlagerung am Genanfang? 1974 endeckten die Wissenschaftler Lynn Dalgarno und John Shine eine spezifische Sequenz der mRNA vor dem eigentlich für das Protein codierenden mRNA-Abschnitts. Diese Sequenz bindet komplementär an die ribosomale RNA (rRNA) der kleinen Untereinheit des Ribosoms. Diese Wechselwirkung sichert, dass das Startcodon exakt in der P-Stelle liegt.
![]() Die Bindungsstellen des Ribosoms
Die Bindungsstellen des Ribosoms
- A - Stelle: Akzeptorstelle oder "Eingang"
- P - Stelle: Peptidyl - tRNA - Stelle, die wachsende Peptid - Kette ist hier gebunden
- E - Stelle: Exit - Stelle, Austrittsstelle oder "Ausgang"
- IF-3 bindet an der kleinen Untereinheit des Ribosoms.
- Dadurch lagert sich kleine Untereinheit des Ribosoms an die mRNA an. Dabei bindet die Ribosomenbindungstelle der mRNA komplementär an die kleine ribosomale RNA (rRNA, kleine Untereinheit des Ribosoms).
- IF-1 lagert sich an und blockiert die A-Stelle
- IF-2 geht eine Verbindung mit der tRNA ein und bewirkt, dass sich die mit fMethion (fMet) beladene Initiator-tRNA an der P-Stelle komplementär zum Startcodon bindet (A-Stelle ist blockiert).
- Dadurch wird IF-3 freigesetzt.
- Danach bindet die große Ribosom - Untereinheit und durch die Hydrolyse von GTP in GDP +Pi wird IF-2 und IF1 freigesetzt.
- Das Ribosom ist jetzt aktiv und der Ablesevorgang (Elongation) kann beginnen.
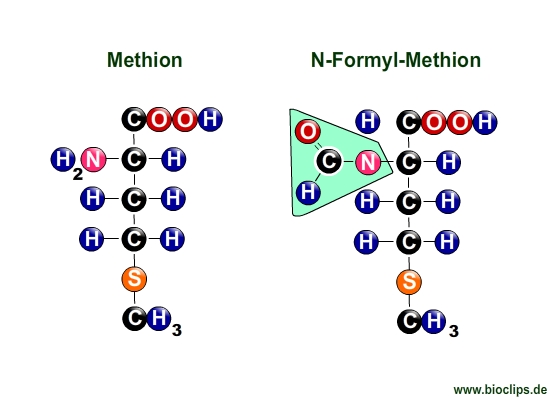
![]() Die Initiator-tRNA (N-Formylmethionyl-tRNA, kurz fMet-tRNA)
Die Initiator-tRNA (N-Formylmethionyl-tRNA, kurz fMet-tRNA)
Bei der ersten t-RNA, die sich am Startcodon anlagert, handelt es sich um eine besondere Form der tRNA. Die sogenannte Initiator-tRNA ist mit N-Formyl-Methion (Formyl-, auch Aldehyd-Gruppe) beladen und nicht mit Methion, welches nach der Initiation im Protein verbaut werden kann.
- Die fMet-tRNA ist die einzige tRNA, die an der P-Stelle angelagert wird.
- Aufgrund der Formyl-Gruppe kann Formyl-Methion keine Peptidbindung ausbilden.
- An ein AUG-Codon innerhalb eines Gens wird eine mit Methion beladene tRNA über die A-Stelle gebunden, so dass dadurch eine Peptidbindung möglich ist.
Elongation - der Ablesevorgang
Elongationsfaktor (EF-Tu; Tu = thermo unstable)
Bei diesem Faktor handelt es sich um ein G-Protein, das wichtige Aufgaben erfüllt:
- Es schützt die Verbindung zwischen tRNA und Aminosäure.
- Durch eine Wechselwirkung mit der großen Untereinheit des Ribosoms sorgt das Protein für eine Anlagerung an der A-Stelle.
- Nur bei einer korrekten komplementären Codon-Anticodon-Bindung kommt es zur Freisetzung von EF-Tu-GTP. GTP reagiert dabei zu GDP+Pi (Hydrolyse). Diese Reaktion kann nur stattfinden, wenn eine korrekte Bindung vorliegt. Bei einer nicht korrekten Bindung kann sich EF-Tu nicht von der tRNA lösen, so dass dadurch der Einbau der richtigen Aminosäure gewährleistet wird.
- EF-TU kann mit der fMet-tRNA keine Verbindungen eingehen, so dass innerhalb des Gens am AUG-Codon kein fMet. eingebaut wird.
Elongationsfaktor EF-G (G - Protein)
Der EF-G ist ebenfalls ein G-Protein, das folgende Aufgaben hat:
- Durch Hydrolyse von GTP kommt es zu einer räumlichen Strukturänderung im Protein. Dadurch werden die in A- und P-Stelle gebundenen tRNA-Moleküle in Richtung E-Stelle "geschubst".
- Die Elongation beginnt, indem EF-Tu-GTP mit der tRNA eine Verbindung eingeht und diese zur A-Stelle leitet.
- Ist die A-Stelle komplementär besetzt, bildet sich zwischen der Aminosäure in der P-Stelle und der Aminosäure in der A-Stelle eine Peptidbindung (Ribozymwirkung) aus.
- Durch die Ausbildung der Peptidbindung wird die Aminosäure (Peptid) von der A-Stelle auf die tRNA der A-Stelle übertragen.
- Nun bindet der EF-G-GTP Komplex am Ribosom und verschiebt die große Untereinheit des Ribosoms um ein Codon.
- Daraufhin bewegt sich die kleine Untereinheit des Ribosom ebenfalls um ein Codon.
- Dadurch gelangt die entladene tRNA zur E-Stelle. Die A-Stelle ist nun wieder frei und kann durch eine beladene tRNA besetzt werden.
- Daraufhin wiederholt sich der gesamte Vorgang bis zum Erreichen des Stopp-Codons.
![]() Das Ribosom ist auch ein Enzym - nämlich ein Ribozym
Das Ribosom ist auch ein Enzym - nämlich ein Ribozym
Das Ribosom katalysiert die Ausbildung der Peptidbindung zwischen zwei Aminosäuren. Enzymatisch wirksam ist die große Untereinheit des Ribosoms, die als Peptidyltransferase wirkt. Hierbei handelt es sich um ein sogenanntes Ribozym, das ohne Proteinbeteiligung enzymatisch aktiv ist.
Termination - der Synthesestopp
Freisetzungsfaktoren - RF-1 oder RF-2 (release factors) RF1lagern sich in der A-Stelle am Stoppcodon an, wobei RF-1 sich an die Stoppcodone UAG und UAA und RF2 an UAA und UGA anlagert.
Freisetzungsfaktor - RF-3 bewirkt die Freisetzung von RF-1 oder RF-2.
Ribosom-Recycling-Faktor - RRF spaltet das Ribosom in seine Untereinheiten.
![]() Der Elongationsfaktor EF-G-GTP hat hier die gleiche Aufgabe wie bei der Elongation.
Der Elongationsfaktor EF-G-GTP hat hier die gleiche Aufgabe wie bei der Elongation.
- In der A-Stelle befindet sich das Stopp-Codon.
- Da es keine komplementäre tRNA gibt, hält das Ribosom an und es bindet RF-1.
- RF-1 aktiviert die Peptidyltransferase, wodurch das fertige Protein an der P-Stelle freigesetzt wird.
- Durch RF-3 wird RF-1 freigesetzt.
- RRF und EF-G-GTP (GTP - GDP + Pi) besetzen die A-Stelle und bewirken die Aufspaltung des Ribosoms in seine Untereinheiten.
- IF-3 entfernt die entladene tRNA und die mRNA von der kleinen ribosomalen Einheit.
Zusammenfassung - Translation
Die Transkription beginnt mit der Initiation. Verschiedene Initiationsproteine kontrollieren die Verknüpfung der mRNA- mit dem Ribosom. Eine spezifische Initiator-tRNA bindet an der P-Stelle. Das Ribosom ist bereit für den Ablesevorgang – Elongation.
Die Elongation ist der eigentliche Ablesevorgang, der zur Synthese des Proteins führt. Nach der Anlagerung beladener t-RNA´s werden die Aminosäuren miteinander verknüpft. Hier wirkt das Ribosom als Ribozym. Verschiedene Elongationsfaktoren kontrollieren die Anlagerung der tRNA´s sowie die Verlagerung der Ribosom-Untereinheiten.
Die Termination ist der Vorgang, der die Translation bei Erreichen des Stoppcodons beendet. Verschiedene Proteine halten das Ribosom an, trennen das fertige Protein sowie die mRNA vom Ribosom, um dann das Ribosom in seine Untereinheiten zu zerlegen.