DNA-Sequenzierung - Kettenabbruchmethode nach SANGER
1975 entwickelte Frederick Sanger ein enzymatisches Analyseverfahren zur Bestimmung der Basenabfolge in DNA – Molekülen. Frederick Sanger war damals in der Lage die Reihenfolge von 5 Basen in der Woche zu bestimmen. In Bezug auf die Analyse des menschlichen Genoms mit 3,5 x 1012 Basenpaaren ein schier unlösbare Aufgabe. Genome von Pflanzen und selbst niedere Organismen können noch größere Genome enthalten. So hat Weizen eine Genom mit 1,6 x1013 und eine Amöbenart 1,2 x 1014 Basenpaare. Heute kann man DNA – Stränge von 1000 bp in verhältnismäßig kurzer Zeit auslesen.
Worum gehts?
Die Kettenabbruchmethode ist ein Verfahren, um die Reihenfolge der Nucleotidbasen in der DNA zu bestimmen.
Funktionsprinzip
- Durch den Abbruch der Replikation an genau definierten Stellen (z.B.: nach der Nucleotidbase mit Adenin) lassen sich DNA-Fragmente unterschiedlicher Länge herstellen.
- Der Längenunterschied der gewonnenen "DNA-Schnipsel" beträgt 1 Nucleotid.
- Diese werden mittels Gelelektrophorese aufgetrennt.
Wie der Abbruch erzeugt wird
- Der Abbruch erfolgt durch den Einbau veränderter Nucleotide, die eine Anlagerung nachfolgender Nucleotide nicht zulassen.
- Dem modifizierten ddNTP fehlt am 3´-Ende die Hydroxylgruppe (-OH), ein Nucleotid kann sich nicht mehr anlagern, es kommt zu Abbruch (Termination)
- Im Versuchsansatz mit Adenin - ddNTP wird immer nach Adenin die Replikation abgebrochen.
- Für die anderen Nucleotidbasen muss ein entsprechender Versuchsansatz angefertigt werden.
- Damit erhält man 4 Proben mit Abbruch nach Adenin, Abbruch nach Thymin ...
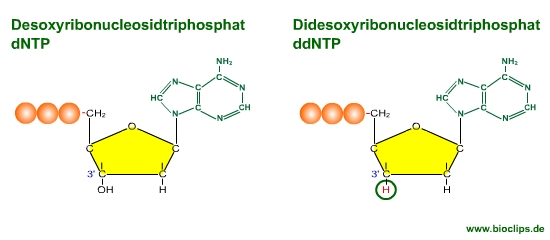
Die Abbildung zeigt die ddNTP für Adenin.
Wie der Kettenabbruch z.B. bei Adenin funktioniert
Der Versuchsansatz für Adenin enthält:
- die zu untersuchende DNA, die denaturiert als Einzelstrang (Matrizen-DNA) vorliegt
- DNA-Polymerase
- Primer (Startbereich für Polymerase)
- die Primer sind radioaktiv markiert, um im Anschluss die DNA-Fragmente über einen Röntgenfilm sichtbar zu machen
- modifizierte ddNTP, die dafür sorgen, dass es zum Kettenabbruch kommt
- das Verhältnis von dNTP zu ddNTP-Adenin beträgt 200:1
![]() "Müssten sich alle DNA-Fragmente der 4 Proben der Größe nach aufstellen, dann würden sie sich um eine Nucleotidbase unterscheiden."
"Müssten sich alle DNA-Fragmente der 4 Proben der Größe nach aufstellen, dann würden sie sich um eine Nucleotidbase unterscheiden."
Die in der Animation dargestellte Synthese läuft nicht nacheinander, sondern parallel ab.
Verlauf
Denaturierung und Neutralisation
- die Denaturierung der Doppelstrang DNA ist Voraussetzung für die Anlagerung der Primer
- das Erzeugen von DNA-Einzelsträngen durch Temperaturerhöhung wirkt nur kurzfristig und reicht für eine vollständige Denaturierung nicht aus
- die Zugabe stark alkalischer Natronlauge (NaOH) bewirkt eine vollständige Denaturierung
Primerhybridisierung
- ein geordneter Synthesestart erfolgt, wenn die DNA-Einzelstränge in linearer, einzelsträngiger Form vorliegen
- die Primer lagern sich an der vorgesehenen Stelle an (Hybridisierung)
- die Geschwindigkeit der Hybridisierung ist abhängig von Länge, Größe und Konzentration des Primers.
- in der Regel ergeben sich daraus Hybridisierungszeiten von 3-5 Sekunden
Strangsynthese
- es erfolgt der Ablese- und Synthesevorgang durch die DNA-Polymerase ähnlich der Replikation
- die dNTPs werden fortlaufend eingebaut, bis sich zufällig ein modifiziertes Nucleotid (ddNTP) anlagert
- dann kommt es zum Syntheseabbruch (Kettenabbruch)
- als Ergebnis erhält man einen DNA-Strang, der nur teilweise aus einem Doppelstrang besteht
Finale Denaturierung
- für eine exakte Größenbestimmung der neu synthetisierten DNA-Fragmente unterschiedlicher Länge müssen diese vom Matrizenstrang getrennt werden
- das erreicht man durch eine Denaturierung der Doppelstrangbereiche
- Hitzebehandlung und Harnstoff, der das Ausbilden von Wasserstoffbrücken verhindert, sorgen für eine Denaturierung
Anmerkung
Die Sanger-Kettenabbruchmethode kann nur für Sequenzen von 200 bis 400 Nucleotide in einer Einzelreaktion benutzt werden. Man muss somit für 1000 Basenpaare verschiedene Läufe durchführen und dann überlappende Sequenzen zusammenpuzzeln.
Auftrennung und Darstellung
In einer Gelelektrophorese werden die DNA-Fragmente ihrer Größe nach aufgetrennt und durch die radioaktive Markierung auf einem Röntgenfilm (Autoradiogramm) sichtbar gemacht.
Die Animation simuliert die Auftrennung der Einzelstrang - Fragmente. Damit lässt sich die Nucleotidbasensequenz bestimmen.
Auswertung
Das Auslesen der DNA-Sequenz beginnt mit dem kürzesten Fragment.
Dabei handelt es sich um den ersten Abbruch nach dem Primer. Es ist dem 5´-Ende des Primers am nächsten, damit ist die Ausleserichtung festgelegt.
Die Nucleotidbasenproben werden hintereinander ausgelesen (siehe Film).
Dem Ausleseergebnis müssen nun noch die komplementären Basen zugeordnet werden.
Bedeutung
Die Nucleotidbasensequenz von Genen zu kennen, kann wichtig sein...
- um die Aminosäuresequenz eines Proteins abzuleiten;
- um die exakte Sequenz eines Gens zu bestimmen;
- um die regulatorischen Elemente zu bestimmen;
- um Unterschiede in Genen zu identifizieren;
- um genetische Mutationen (Polymorphismen) zu bestimmen.